Biologia molecolare applicata in micologia
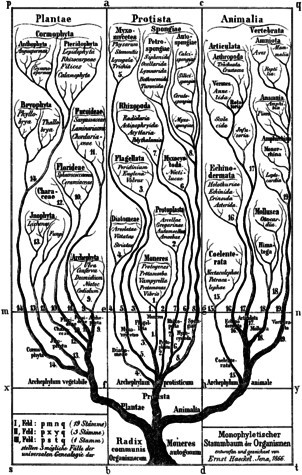
Ernst Haeckel, 1866: l’Ontogenesi ricapitola la Filogenesi.
Orlando Petrini, febbraio 2017
Indice
PROGRAMMA.............................................................................................................................. 2
MATERIALE NECESSARIO.............................................................................................................. 2
ALCUNE DEFINIZIONI.................................................................................................................... 3
COME DEFINIRE UN GENERE?....................................................................................................... 7
LETTERATURA RACCOMANDATA.................................................................................................. 8
SOFTWARE PER ANALISI FILOGENETICHE....................................................................................... 8
Programma
09:30 – 11:00 Biologia molecolare e genetica
DNA e sequenze
L’orologio molecolare e le mutazioni
Genomica e proteomica
Genomica e proteomica in micologia Filogenia e morfologia
11:30 – 12:30 Tassonomia polifasica
Introduzione alla filogenia
14:30 – 16:30 Analisi filogenetiche e fenetiche
Analisi fenetiche - analisi filogenetiche
Dendrogrammi - cladogrammi - alberi filogenetici
Indici di similarità
Analisi filogenetiche - regole di base
Lettura di un albero filogenetico
Tecniche di base per le analisi filogenetiche
Esercizi di comprensione
Come definire un genere
16:30 – 17:30 Biologia molecolare e tassonomia – domande e risposte
Materiale necessario:
Dispense e pubblicazioni distribuite
Materiale per prendere appunti
Alcune definizioni*
|
Adattamento
|
Adeguamento (morfologico e fisiologico) degli esseri viventi alle condizioni ambientali.
|
|
Albero filogenetico
|
Diagramma che mostra le relazioni fondamentali di discendenza comune di gruppi tassonomici di organismi.
La rappresentazione delle relazioni in questa forma è tipica della visione evoluzionistica espressa secondo i suoi concetti iniziali, secondo la quale lo sviluppo delle forme di vita è avvenuto a partire da un progenitore comune (il tronco o la base dell'albero, altrimenti detta radice), il quale ha dato origine per speciazione a diverse linee di discendenza, fino ad arrivare alle specie attualmente esistenti (le cime dell'albero, altrimenti dette ramificazioni terminali). In un albero filogenetico, ciascun nodo (o biforcazione) rappresenta l'antenato comune più recente dei soggetti che si trovano ai nodi successivi e la lunghezza delle ramificazioni può essere correlata al tempo o ai cambiamenti genetici che intercorrono tra di essi.
|
|
Barcoding
|
Metodica molecolare per l’identificazione di organismi, Basata sull’analisi della variabilità di un marcatore molecolare (funghi: spesso ITS, ma anche calmodulina, beta-tubulina, ecc.).
|
|
Bayes (statistica Bayesiana)
|
La statistica Bayesiana è un campo della statistica in cui l'evidenza su uno stato vero del mondo è espressa in termini di gradi di credibilità o più specificamente di probabilità Bayesiana.
|
|
Bayesiana (inferenza)
|
L’inferenza Bayesiana utilizza probabilità antecedenti e
posteriori per misurare la qualità della filogenia ottenuta.
|
|
Bootstrap
|
Il bootstrap è una tecnica statistica di ricampionamento con reimmissione per approssimare la distribuzione campionaria di una statistica. Permette perciò di approssimare media e varianza di uno stimatore, costruire intervalli di confidenza e calcolare i valori p (probabilità) di test quando, in particolare, non si conosce la distribuzione della statistica di interesse.
|
|
Clade
|
Gruppo di organismi costituito da un antenato singolo comune e da tutti i discendenti di quell'antenato. Qualsiasi gruppo che corrisponde alla definizione viene considerato monofiletico e può essere rappresentato o da un'analisi
filogenetica o da un cladogramma.
|
* In parte tratte da Wikipedia e modificate quando necessario.
| |
Il termine deriva dal greco antico κλάδος (kládos = ramo), ed
è stato coniato nel 1958 dal biologo Julian Huxley.
Se un clade è considerato solidamente attestato in svariate analisi, usando differenti insiemi di dati, può entrare a far parte della tassonomia e venire considerato un taxon. Non tutti i taxa considerati tali sono cladi. I rettili, per esempio, sono un gruppo parafiletico perché non includono gli uccelli, che sono considerati discendenti di un gruppo di rettili, gli arcosauri.
|
|
Cladogramma
|
Un diagramma rappresentante delle parentele. Rappresenta solo i legami di parentela tra le specie incluse nel cladogramma. Il tempo non è incluso nell'analisi né nel grafico e il grado di parentela è tanto più grande tanti più nodi due specie condividono. Le forme basali non sono antenate delle derivate, ma solo più lontane da queste ultime in termini di parentela. Il fattore tempo e le relazioni antenato- discendente non sono oggetto dell'analisi che produce il cladogramma: essi sono studiati in altri tipi di analisi (vedi albero filogenetico).
|
|
Classificazione
|
Il raggruppare delle unità tassonomiche in base alla loro similarità/dissimilarità.
|
|
Cluster (analisi di)
|
Procedura che permette di rappresentare graficamente dei raggruppamenti.
|
|
Codice genetico
|
Regole attraverso le quali viene tradotta l'informazione codificata nel materiale genetico (DNA) in proteine. Quasi tutti gli esseri viventi usano il medesimo codice genetico.
|
|
Coevoluzione
|
Insieme delle modificazioni adattive che si manifestano nel tempo in specie legate tra loro da rapporti di interdipendenza (ad es. simbiosi).
|
|
Dendrogramma
|
Strumento grafico per la visualizzazione dei coefficienti di similarità quantificato nel processo di “raggruppamento”. Nelle tecniche di clustering, il dendrogramma è utilizzato per fornire una rappresentazione grafica del processo di raggruppamento delle istanze (o unità statistiche, o records, o elementi dell'insieme). Il dendrogramma dà una rappresentazione fenetica delle differenze, senza necessariamente fornire inferenze sul grado di parentela effettiva di due o più organismi.
|
|
Fenetica (analisi)
|
Si basa sulle similarità (dissimilarità) tra organismi e spiega quanto simili tra di loro siano due o più organismi. Rappresentazione grafica: il dendrogramma.
|
|
Filogenesi
|
Lo studio della storia evolutiva degli organismi viventi.
L’analisi filogenetica molecolare si basa sulla distanza
genetica tra coppie di sequenze (DNA o proteine) e spiega
|
| |
quanto vicini tra di loro, da un punto di vista evolutivo, siano due o più organismi.
Rappresentazione grafica: l’albero filogenetico (ev. cladogramma), che contiene i tempi e gli schemi temporali dei processi di divergenza.
|
|
Genoma
|
La totalità del DNA aploide di una cellula
|
|
Filogenesi molecolare
|
Tecnica per lo studio della storia evolutiva degli organismi viventi, basata sull’analisi delle sequenze molecolari (DNA o amminoacidi delle proteine).
|
|
Identificazione
|
Il processo di denominazione di un organismo.
|
|
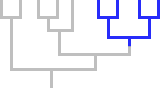
Monofiletico
|
Un insieme che comprende almeno due taxa, il loro antenato comune e tutti i suoi discendenti.
|
|
Nodi
|
In un albero tassonomiche.
|
filogenetico
|
rappresentano
|
le
|
unità
|
|
Ontogenesi
|
L'insieme dei processi mediante i quali si compie lo sviluppo biologico di un organismo vivente dall'embrione allo stadio adulto: dipende sia dal genoma, che caratterizza l'organismo, sia dall'ambiente nel quale il processo si svolge.
|
|
Ordinazione
|
Il processo di ordinare un gruppo di punti nello spazio.
|
|
Orologio molecolare
|
Parte dall’ipotesi che mutazioni casuali, con le quali i geni si evolvono, si verifichino con frequenze pressoché costanti nel tempo. Diventa quindi possibile stimare il tempo trascorso dal momento in cui si è verificata la divergenza tra due specie che discendono dallo stesso antenato comune valutando il numero delle differenze presenti in sequenze di DNA correlate o nelle corrispettive proteine.
|
|
Outgroup
|
Gruppo “esterno” alla filogenesi studiata, usato per produrre
alberi filogenetici radicati.
|
|
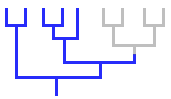
Parafiletico
|
Un gruppo non naturale, incompleto, al cui interno è presente l'antenato comune ma non tutti i suoi discendenti.
|
|
Parsimonia (criterio di)
|
Tra diverse possibili spiegazioni di un fenomeno osservato, preferisce quella che richiede il numero minore di assunzioni/cambiamenti. Secondo questo criterio, una filogenia è ottimale (o più parsimoniosa) se la somma dei pesi degli archi di ogni percorso da un taxon all’altro è minima.
|
|
PCR
|
Reazione a catena della polimerasi (“Polymerase Chain Reaction”). Moltiplicazione (amplificazione) di frammenti di
|
| |
acidi nucleici di cui si conoscono le sequenze iniziali e terminali. Per una PCR sono necessari dei cosiddetti primer (“innesco”), filamenti di acido nucleico (18-20 basi) che serve come punto d’innesco per la replicazione del DNA. La PCR produce sequenze di geni ben definiti che possono essere usate per lavori di filogenetica.
|
|
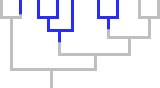
Polifiletico
|
Un gruppo che comprende dei discendenti senza includere gli antenati comuni (perché questi non ci sono o non hanno le stesse caratteristiche che uniscono i discedenti).
|
|
Proteoma
|
Insieme delle proteine di un organismo
|
|
Rami
|
In un albero filogenetico uniscono i nodi e rappresentano le distanze tra due nodi
|
|
Radice (di un albero filogenetico)
|
Un albero radicato (in inglese rooted) viene visualizzato tramite un grafo diretto, una struttura ad albero che si sviluppa a partire da un unico nodo, rappresentante il più recente antenato comune delle forme di vita che si trovano alle estremità dell'albero. In questo modo, un albero filogenetico radicato è in grado di fornire informazioni sia sulla correlazione genetica esistente tra gli organismi presenti sulle sue ramificazioni, sia sui rapporti evolutivi che intercorrono tra gli stessi (vedi figura 1).
Un albero non radicato (in inglese unrooted), al contrario, illustra le relazioni genetiche che intercorrono tra gli organismi che si trovano ai suoi apici, ma non fornisce alcuna informazione in merito alla loro evoluzione (vedi figura 2).
|
|
Similarità (misure)
|
Indici (spesso arbitrari) della somiglianza tra due unità tassonomiche (non necessariamente organismi). Sono usati per la costruzione di alberi fenetici.
· Similarità simmetrica: assenza di un carattere è informativa;
· Similarità asimmetrica: l’assenza non è un dato certo.
|
|
Sistematica
|
Ramo della biologia che si occupa della classificazione. Spesso messa in sinonimia con tassonomia. Vi è però una sottile ma importante differenza tra i due termini:
· La sistematica studia e stabilisce le relazioni tra gli organismi per permetterne la classificazione;
· La tassonomia descrive, dà il nome e ordina gli organismi: fornisce quindi i mezzi necessari all’identificazione degli organismi.
|
|
Speciazione
|
Processo di formazione e differenziazione delle specie viventi.
|
|
Tassonomia polifasica
|
Sistema di classificazione che combina e sintetizza tutta
l’informazione disponibile sugli organismi studiati. Produce
|
| |
quindi una tassonomia completa, che necessariamente diventa multifattoriale. In micologia questa filosofia ha fatto il suo ingresso negli anni novanta.
|
|
Topologia
|
Struttura generale di un albero filogenetico.
|
|
Trascrizione
|
una sequenza di DNA, chiamata gene strutturale, è usata come stampo per la creazione di un filamento complementare di RNA (acido ribonucleico).
|
|
Traduzione
|
Una sequenza di RNA si compone di gruppi non sovrapposti di tre basi ciascuno, chiamati codoni. Ad ogni codone corrisponde uno specifico amminoacido. La sequenza di codoni (o triplette di basi) è tradotta in una sequenza di amminoacidi. Esistono 64 codoni.
|
|
Verosimiglianza (criterio di)
|
Tra diverse possibili spiegazioni di un fenomeno osservato, bisognerebbe preferire quella che ha una probabilità più alta di occorrere. Secondo questo criterio una filogenia è ottimale (o la più probabile) se ha la più alta probabilità di spiegare le
relazioni tra i taxa osservati.
|
Come definire un genere?
Riassunto del lavoro di Vellinga et al. Six simple guidelines for introducing new genera of fungi.
IMA Fungus. 2015;6(2):65-8.
La descrizione di un nuovo genere dovrebbe sottostare a dei criteri precisi, miranti a definire
all’interno della comunità micologica un concetto di genere universalmente accettato.
A questo proposito si raccomanda di seguire i sei criteri qui elencati.
1. Criterio di reciproca monofilia
Tutti i generi di un gruppo dovrebbero essere monofiletici, sia nel gruppo da cui il nuovo
genere è separato sia in quello cui il nuovo genere sarà assegnato
2. Concetto di ampietà
L’albero filogenetico usato per stabilire il genere deve contenere
- Un grosso numero di specie
- Una distribuzione geografica di taxa abbastanza grande
- Tipi di tutte le specie dei generi studiati
3. Concetto di stabilità statistica
I rami dell’albero filogenetico devono avere un buon supporto statistico
4. “In dubio contra reum”
Altre opzioni, oltre alla definizione di un nuovo genere, devono essere esaminate e
ponderate: anche in un albero filogenetico robusto si possono vedere diverse possibilità
di interpretazione. [A questo proposito io suggerisco anche un uso ponderato della
tassonomia polifasica!]
5. Criterio genetico
L’evidenza filogenetica non deve basarsi sulle sequenze di un solo gene (idealmente
almeno 3 geni codificanti proteine oltre al “tipico” ITS)
6. Criterio del “peer-review”
Il lavoro deve essere pubblicato su una rivista “peer-reviewed” per permettere una critica
da parte di esperti del gruppo (“controllo di qualità)
Letteratura raccomandata
- Albertini E. filogenesi molecolare.(http://www.emidioalbertini.com/pdf/Filogenesi%20molecolare.pdf)
- Dentinger BT, Ammirati JF, Both EE, Desjardin DE, Halling RE, Henkel TW, et Molecular phylogenetics of porcini mushrooms (Boletus section Boletus). Molecular Phylogenetics and Evolution. 2010;57(3):1276-92.
- Pinnoi A, Phongpaichit P, Jeewon R, Tang A, Hyde K, Jones E. Phylogenetic relationships of Astrocystis eleiodoxae nov. (Xylariaceae). Mycosphere. 2010;1:1-9.
- Tang AM, Jeewon R, Hyde KD. Phylogenetic relationships of Nemania plumbea nov. and related taxa based on ribosomal ITS and RPB2 sequences. Mycological Research. 2007;111(Pt 4):392-402.
- Vellinga EC, Kuyper TW, Ammirati J, Desjardin DE, Halling RE, Justo A, et al. Six simple guidelines for introducing new genera of fungi. IMA Fungus. 2015;6(2):65-8.
Software per analisi filogenetiche
- BEAST – Bayesian estimation (http://beast.bio.ed.ac.uk/Main_Page).
- FigTree (http://tree.bio.ed.ac.uk) per la grafica
- MEGA-7 (https://www.megasoftware.net/megabeta.php)
- MrBayes: Bayesian Inference of Phylogeny (http://mrbayes.sourceforge.net/ )
- SeaView (https://doua.prabi.fr/software/seaview)
- Geneious (https://www.geneious.com/)
Cos'è giusto e cosa sbagliato? - Esercizio 1
FIG. 1. Dendrogramma “Neighbor joining” basato su 486 bp (paia di nucleotidi) della regione genica ITS1-2 (Kimura-2, bootstrap test con 500 repliche). F: Francia; CH: Svizzera; D: Germania; JAP: Giappone; IND: India; ESP: Spagna; nd: origine sconosciuta.
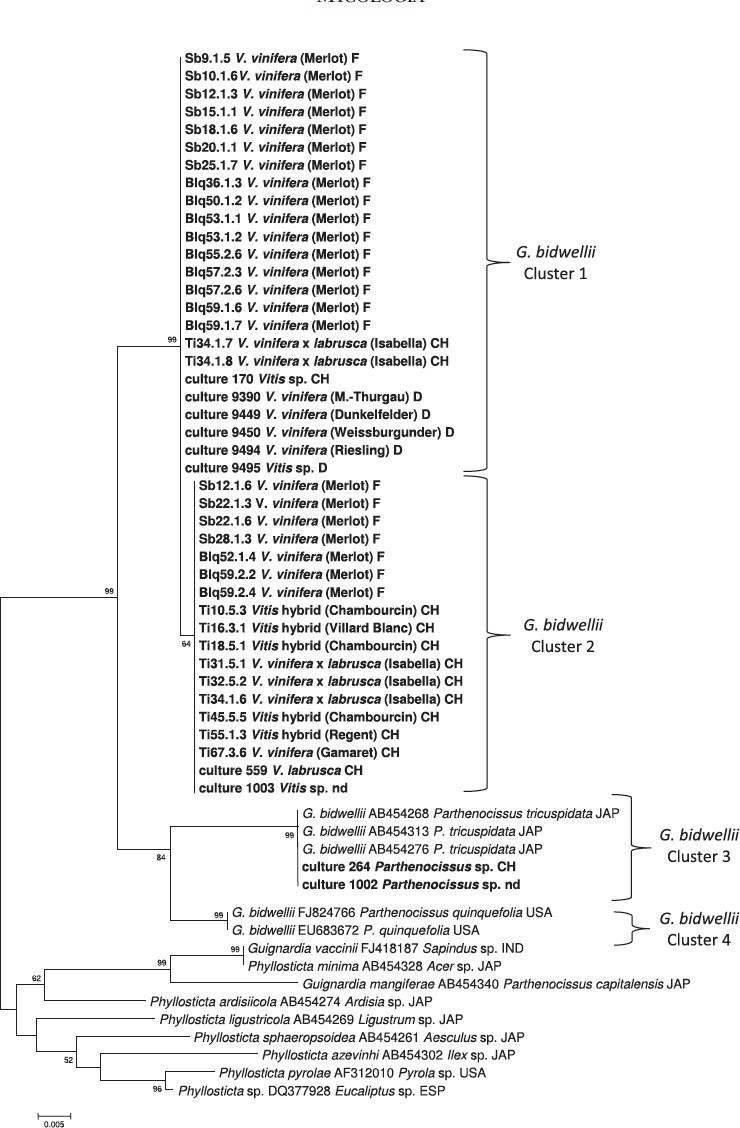
Bibliografia:
Wicht B, Petrini O, Jermini M, Gessler C, Broggini GA. Molecular, proteomic and morphological characterization of the ascomycete Guignardia bidwellii, agent of grape black rot: a polyphasic approach to fungal identification. Mycologia. 2012;104(5):1036-45.
Fig. 2. a) dendrogramma dell’analisi filogenetica eseguita usando le sequenze parziali del gene ITS. b) albero filogenetico costruito usando le sequenze del gene tef1. c) albero filogenetico costruito con le similarità ottenuto tramite analisi MALDI-TOF MS.
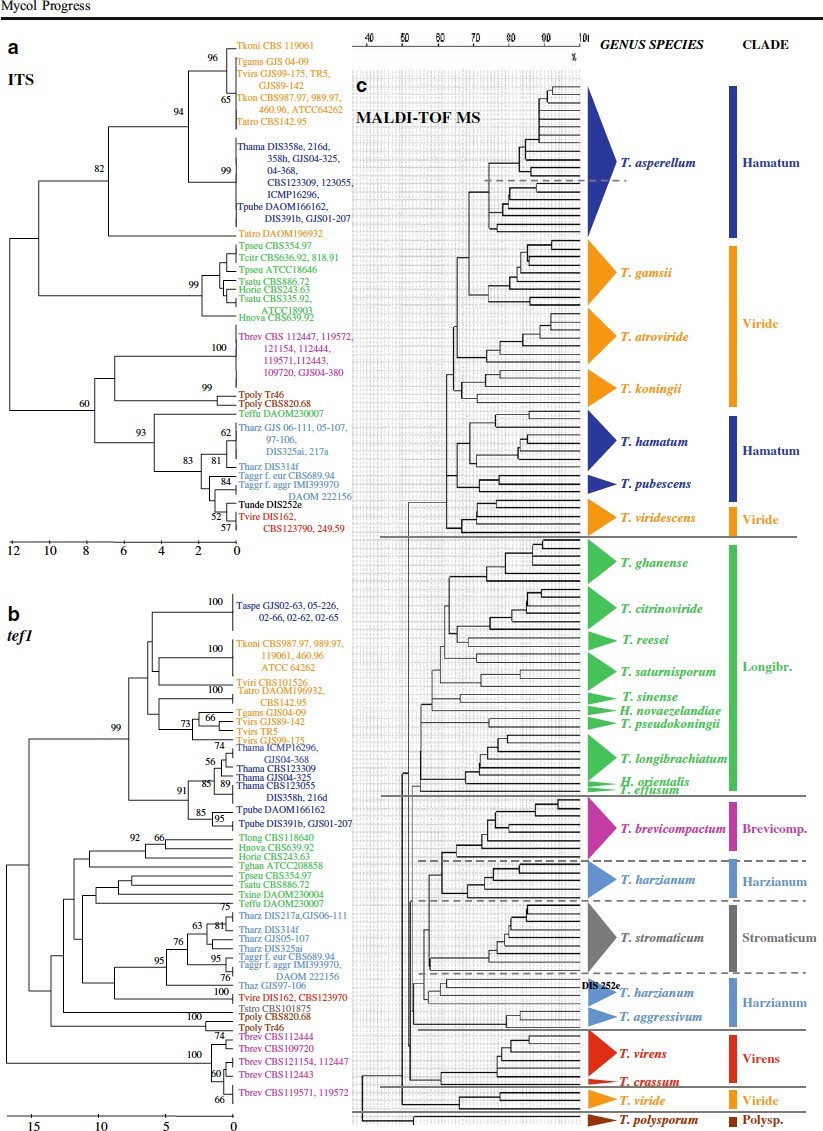
Bibliografia:
De Respinis S, Vogel G, Benagli C, Tonolla M, Petrini O, Samuels G. MALDI-TOF MS of Trichoderma: a model system for the identification of microfungi. Mycological Progress. 2010;9(1):79-100.
Fig. 3. a) Dendrogramma dell’analisi filogenetica eseguita usando le sequenze parziali del gene ITS di collezioni di Tetracladium spp. raccolte in Francia e Svizzera. TEMA: Tetracladium marchalianum.
| |
Sì
|
Non
menzionato
|
Commenti
|
|
Numero sufficiente di campioni?
|
|
|
|
|
Organismi vicini e gruppi esterni (outgroup) presi in considerazione?
|
|
|
|
|
Outgroup appropriato / discusso?
|
|
|
|
|
Differenze geografiche considerate?
|
|
|
|
|
Differenze ecologiche considerate?
|
|
|
|
|
Geni e/o proteine considerati – giustificazione a seconda del livello e del gruppo tassonomico studiato?
|
|
|
|
|
Statistiche presentate / discusse?
|
|
|
|
|
Differenze morfologiche considerate e discusse?
|
|
|
|
Sei semplici linee guida per introdurre nuovi Generi di funghi
Else C. Vellinga, Thomas W. Kuyper, Joe Ammirati, Dennis E. Desjardin, Roy E. Halling, Alfredo Justo, Thomas laessoe, Teresa Lebel, D. Jean Lodge, P. Brandon Matheny, Andrew S. Methuen, Pierre-Arthur Moreau, Gregory M. Mueller, Machiel E. Noordeloos, Jorinde Nuytinck, Clark L.
Ovrebo, and Annemieke Verbeken
[Vellinga et al. Six simple guidelines for introducing new genera of fungi. IMA Fungus. 2015;6(2):65-8.]
Traduzione di Giovanni Consiglio
INTRODUZIONE
Solo nel 2014 e nei primi sei mesi del 2015 sono stati proposti più di 20 nuovi Generi nelle Boletaceae. La maggior parte di questi nuovi nomi generici comprendono specie che sono presenti in Nord America e in Europa e che per lungo tempo sono state chiamate “Boletus”.
I numeri per le Agaricales sono confrontabili: abbiamo contato circa 25 nuovi nomi di Genere pubblicati nello stesso periodo di tempo, la maggior parte dei quali a sporata bianca, con sei nuovi Generi per specie che eravamo soliti chiamare Clitocybe, cinque nuovi Generi nelle Lyophyllaceae e tre nelle Psathyrellacae. Il Genere di gran lunga il più vasto, Cortinarius, non è stato influenzato. Invece, probabilmente 6-7 Generi sono stati ora inclusi in Cortinarius. Con i molti nuovi nomi generici introdotti per specie ben note, giunge come una sorpresa anche più grande notare che sono stati descritti pochissimi nuovi Generi basati su specie scoperte recentemente con una combinazione unica di caratteri morfologici. Esempi di questi ultimi sono Cercopemyces crocodilinus delle Rocky Mountains (USA) e Hymenoporus paradoxus della Cina (Baroni et al. 2014, Tkalčec et al. 2015).
Il principio di base per il riconoscimento di un Genere, o di un qualunque rango tassonomico per questa materia, è la monofilia (Henning 1950, 1965). Nel passato ciò è stato estremamente difficile da dimostrare nei funghi, poiché il numero dei caratteri morfologici che potevano essere usati era limitato. Era anche sconosciuto se alcuni dei caratteri che venivano usati, come la formazione di basidiomi sequestrati o gasteroidi, avevano una bassa evolvibilità, giustificando il riconoscimento come Generi separati, oppure un’alta evolvibilità, che allora minimizza la sua rilevanza in un contesto filogenetico.
L’inondazione di questi recenti nuovi nomi generici è stata stimolata principalmente dalla ricerca filogenetica-molecolare e dai risultanti alberi filogenetici. Ciò ha portato alla rivalutazione di caratteri; in molti casi forme sequestrate e gasteroidi sono state dimostrate possedere
un’evolvibilità maggiore di quella assunta, giustificando l’inclusione di tali Generi in Generi esistenti; per esempio in Suillus (Kretzer & Bruns 1997), Cortinarius (Peintner et al. 2002), Lactarius (Eberhardt & Verbeken 2004, Kirk 2015), Russula (Lebel & Tonkin 2007), Lepiota (Ge & Smith 2013) e Boletus (Nuhn et al. 2013). La lichenizzazione ha avuto una bassa evolvibilità nei basidiomiceti, giustificando il riconoscimento di Lichenomphalia come Genere separato per specie che precedentemente erano poste in tre Generi (Redhead et al. 2002).
Tuttavia, tali analisi filogenetiche hanno risolto i problemi della delimitazione e del riconoscimento dei Generi in una misura inferiore a quanto si fosse sperato. Le analisi hanno dimostrato che parecchi Generi ben noti restano parafiletici, come Boletus (Nuhn et al. 2013, Wu et al. 2014) e Psathyrella (per es. Padamsee et al. 2008). La trattazione dell’argomento della parafilia ha posto allora la comunità micologica su due percorsi piuttosto divergenti. Questi differenti approcci riflettevano le vecchie distinzioni di moltiplicatori e sinonimizzatori. Un percorso era prendere piccoli gruppi monofiletici come basi per i nuovi Generi, senza troppa preoccupazione del resto del Genere originale. Questa è una pratica mostrata chiaramente dalla trattazione di Boletus e Clitocybe. La separazione di un piccolo gruppo monofiletico spesso ha messo in moto una palla di neve separatrice, come in Xerocomellus (Gelardi et al. 2015, Vizzini 2015). Sfortunatamente, il riconoscimento di tali piccoli Generi talvolta ha contribuito alla formazione di Generi parafiletici, come nel caso di Resupinatus che è diventato parafiletico a causa del riconoscimento di R. vetlinianus in un Genere separato (Petersen et al. 2015). Il secondo approccio è stato adottato in Entoloma e Clitopilus (Co-David et al. 2009), dove è stato scelto deliberatamente un concetto largo di Genere [anche se altri autori hanno optato per Generi più piccoli (per es., Largent 1994, Kluting et al. 2014)]. Altri esempi sono Amanita (Justo et al. 2010) e Cortinarius (Peintner et al. 2001) dove specie sequestrate e gasteroidi sono state incluse in Generi con basidiomi prevalentemente agaricoidi. Come dovrebbe essere spiegata questa divergenza è una questione interessante; certamente la tassonomia esistente del gruppo in questione svolge un ruolo importante. Nei boleti, per esempio, molti nomi generici pubblicati precedentemente per Generi ben supportati, come Leccinum e Strobilomyces, erano già disponibili e la proposta di nuovi Generi è stata (conseguentemente) considerata accettabile.
PRINCIPALI PREOCCUPAZIONI
Troviamo inquietanti parecchie delle recenti tendenze nella ricerca tassonomica micologica sui funghi basidiomiceti:
1. In parecchi gruppi, il passaggio da un albero filogenetico a una classificazione è portato agli estremi, dove ogni singolo clado viene riconosciuto come un Genere separato. Ciò non aumenta la comprensione della storia evolutiva del gruppo in questione; gonfia soltanto l’impianto tassonomico. Da una prospettiva filogenetica formale può non rilevare se abbiamo una famiglia (per es., Boletaceae), con più di cento Generi oppure se abbiamo un Genere (per es., Boletus s.l.) con molte unità infrageneriche, denominate o no formalmente. Nel caso dei boleti, l’inclusione di più sequenze da più taxa può impattare la filogenesi, poiché la
risoluzione della filogenesi delle Boletaceae è bassa su molti rami (Nuhn et al. 2013, Wu et al. 2014). Noi auspichiamo fortemente che siano esplorate e discusse differenti opzioni, invece di usare un modello standard nel quale ogni clado monofiletico è trasferito in un Genere.
2. Parecchi dei nuovi Generi vengono eretti basandosi soltanto sull’evidenza filogenetica fornita da uno o due regioni geniche, talvolta solo sequenze nrITS che non portano in sé stesse a filogenie di un livello più alto (Bruns 2001).
3. Sempre più numerose pubblicazioni rapide, on-line, non revisionate da esperti del settore, appaiono senza alcuna evidenza di supporto per i taxa descritti come nuovi.
Pertanto noi abbiamo formulato e presentiamo qui dei criteri con i quali gli studi filogenetici e le pubblicazioni che presentano dati provenienti da quegli studi dovrebbero essere verificati prima di essere accettati.
LINEE GUIDA PROPOSTE
Alcune delle nostre linee guida proposte sono così auto-esplicative e ovvie per noi che ci sembra superfluo presentarle qui; tuttavia, esempi che sono in conflitto con queste raccomandazioni sono sorprendentemente facili da trovare. Gli esempi che presentiamo mostrano come è facile soddisfare questi standard. Ci rendiamo conto che questioni facili, come la perdita dei typi originali o materiale che non fornisce facilmente sequenze DNA possono interferire con la perfezione, ma si dovrebbe almeno tentare e discutere gli insuccessi. Sottolineiamo l’importanza di esplorare differenti opzioni di classificazione e di fornire argomenti per le nuove tassonomie proposte. Gli esempi che presentiamo sono presi dalla letteratura sui funghi basidiomiceti, ma potrebbero ugualmente essere stati scelti dalla letteratura degli ascomiceti. I primi cinque criteri si riferiscono alla scienza di base; i primi due criteri sono ugualmente importanti e noi poniamo meno enfasi sugli altri tre. Tuttavia, va da sé che dovrebbe essere evitata la contraddizione di qualunque di questi criteri. Raccomandiamo prima di tutto che i ricercatori usino queste linee guida ma anche che i revisori e gli editori di riviste tassonomiche usino questi criteri nella loro valutazione dei manoscritti presentati.
Il sesto criterio riguarda il modo in cui i risultati vengono pubblicati e presentati al mondo scientifico.
1. Tutti i Generi riconosciuti dovrebbero essere monofiletici, non solo quello che è il focus dello studio, ma anche il gruppo dal quale viene separato e il gruppo al quale viene aggiunto (criterio della monofilia reciproca).
Esempi:
(i) Quando Macrolepiota fu scisso in un nucleo Macrolepiota monofiletico con M. procera come specie tipo e in un secondo gruppo contenente M. rhacodes, quest’ultima fu trasferita in Chlorophyllum che in sé stesso era monofiletico solo includendo anche
Endoptychum agaricoides (Vellinga et al. 2003) e vennero fatti tutti i necessari cambiamenti nomenclaturali (Vellinga 2002, Vellinga e de Kok 2002).
(ii) Il Genere Porpoloma fu trovato essere altamente polifiletico e separato in quattro Generi (Sánchez-García et al. 2014). Per l’applicazione di nomi ai cladi è stato essenziale sequenziare la specie tipo di Porpoloma (vedi Raccomandazione 2).
Sfortunatamente molti studi non raggiungono questo standard, dato che è molto facile espellere specie da un Genere senza prendere in considerazione la monofilia del gruppo di arrivo. Specialmente nel caso di scarsa risoluzione in una filogenesi, l’erezione di Generi monofiletici molto piccoli creerà sempre più numerose e sempre più disordinate unità parafiletiche, dalle quali può già essere prevista la successiva separazione di un nuovo Genere.
2. La copertura dell’albero filogenetico deve essere larga.
La copertura deve essere larga in termini:
a) del numero di specie - è importante ricordare che un albero filogenetico dà informazioni solo su quei taxa che sono inclusi in esso e che tutte le affermazioni sulle relazioni sono relative e non assolute a meno che non vengano inclusi tutti i taxa;
b) della distribuzione geografica dei taxa - una filogenesi basata su specie delle aree temperate di Nord America ed Europa non è informativa se il gruppo in questione è rappresentato da molte più specie in Africa tropicale, Asia e Australia;
c) i data base dovrebbero includere le specie tipo di tutti i Generi che si stanno includendo, poiché la collocazione della specie tipo decide quale nome usare per un Genere.
Esempi:
(i) È stato dimostrato che il Genere Anamika cade nel mezzo di Hebeloma quando alla filogenesi basata principalmente su specie euroasiatiche sono stati aggiunti campioni dell’Australia (Rees et al. 2013).
(ii) Solo includendo la specie tipo di Pachylepyrium nelle analisi filogenetiche, si è potuta determinare in modo certo la posizione di P. carbonicola e di P. funariophilum (Matheny et al. 2015).
(iii) La posizione di Marasmius sect. Hygrometrici è stata determinata dalle analisi che includono esplicitamente i typi del Genere Marasmius (e quindi della Sezione Marasmius) e della Sezione Hygrometrici (Jenckinson et al. 2014).
(iv) Il tipo di Rubinolubetus si raggruppa sempre con Chalciporus e quindi il Genere è incluso in
Chalciporus (Nuhn et al. 2013).
3. Le ramificazioni degli alberi filogenetici dovrebbero avere un supporto statistico sufficiente e forte.
Un supporto debole (o anche un’assenza di supporto statistico) dei nuovi Generi proposti indica che non possono essere rigettate classificazioni alternative. E così il consiglio è: “in dubio abstine”, quando sei in dubbio, rinuncia a proporre nuovi Generi.
Esempio:
Lenzites warneri occupa una posizione isolata e non supportata all’interno di Trametes s.l.; Welti et al. (2012) rinunciarono a introdurre un nome generico nuovo per questa specie, anche perché essa non ha caratteri morfologici discriminanti. Neanche Justo & Hibbett (2011) l’hanno riconosciuto come un Genere separato, ma l’hanno incluso nel loro concetto di Trametes, che è più largo di quello di Welti et al. (2012).
4. Dovrebbe essere discusso un elenco di opzioni, dovrebbero essere provate differenti opzioni e forniti argomenti per la decisione finale.
Spesso i metodi filogenetici permettono, anche in alberi ben risolti, più d’una classificazione formale con gruppi monofiletici. Pertanto dovrebbero essere presentate e discusse opzioni differenti.
Esempi:
(i) Justo & Hibbett (2014) e Justo (2014) hanno discusso le differenti opzioni e le relative conseguenze per riconoscere dieci o cinque Generi o appena un Genere all’interno di Trametes s.l., optando alla fine per una soluzione monogenerica.
(ii) Halling et al. (2015) hanno provato differenti opzioni per la circoscrizione di Boletellus e per la sua delimitazione rispetto a Heimioporus; questi autori inclusero la specie che forma un grado alla base del nucleo Boletellus all’interno di quel Genere, invece di descrivere nuovi Generi per ciascun clado.
(iii) Buyck et al. (2008) hanno presentato e provato 15 differenti opzioni per la filogenesi dei differenti cladi in Lactarius e Russula, prima di arrivare alla soluzione di spezzare Lactarius in due Generi, Lactarius e Lactifluus e di riconoscere Russula Sez. Ochricompactae come Genere separato, Multifurca.
(iv) Lodge et al. (2014) hanno discusso le opzioni per il riconoscimento di uno o tre Generi per
Gliophorus, Porpolomopsis e Neohygrocybe.
5. Le evidenze filogenetiche devono essere basate su più di un gene, preferibilmente geni che codificano proteine in aggiunta a regioni geniche della replica SSU-ITSU-LSU.
Le differenti regioni geniche che vengono comunemente usate nella classificazione dei basidiomiceti hanno tutte differenti storie evolutive e quindi hanno un differente potere risolvente a differenti livelli. Filogenesi basate solo su sequenze nrITS non solo devono essere affrontate con
una certa dose di scetticismo ma di fatto non dovrebbero più essere accettate come base per nuovi Generi. Questo marcatore è il codice a barre universale per il riconoscimento di specie fungine (Scoch et al. 2012) e svolge questo ruolo generalmente bene a causa di una variazione sufficientemente larga tra le specie correlate con molti, ma non tutti, gruppi fungini; il prezzo che paghiamo per questa accuratezza a livello di specie è che l’nrITS non è allineabile su taxa con relazioni più distanti (Bruns 2001). In passato, è stato dibattuto cosa fosse meglio, avere più taxa/raccolte analizzati o più geni per meno taxa (Greybeal 1998); con minori costi di sequenziazione e computer più veloci, la risposta è più taxa e più geni. Comunque, filogenesi basate su genomi completi sono ancora rare e comprendono solo un piccolo numero di specie (per es., Dentinger et al. 2015); e naturalmente, i confronti dell’intero genoma saranno affrontati con gli stessi argomenti delle filogenesi basate su molti geni.
Esempio:
In un’analisi a tre loci (geni nLSU, nSSU e rpb2) di un vasto intervallo di taxa nel clado tricholomatoide, Sánchez-García et al. (2014) hanno riconosciuto i nuovi Generi Corneriella, Albomagister, Pogonoloma e Pseudotricholoma tutti con pieno supporto statistico.
6. Nella pubblicazione in cui i nuovi taxi vengono proposti, dovrebbero essere incluse tutte le evidenze di supporto e le informazioni di base; e in secondo luogo, questa pubblicazione dovrebbe essere rivista da esperti del settore.
La prima parte di questa linea guida è suggerita dall’apparizione di pubblicazioni molto corte associate a uno dei siti di registrazione di taxa ufficiali, senza alcuna evidenza di supporto né illustrazioni, talvolta con un rimando a un altro spesso personale sito web dove si possono trovare le informazioni di supporto (per es., un albero filogenetico). La comunicazione dei risultati della scienza non è un evento di una sola volta. Un principio basilare della scienza è che i risultati siano verificabili da altri. Se in futuro i ricercatori non possono risalire all’intero set di dati e di informazioni, questo principio non può essere applicato. Questa situazione è la stessa degli studi sperimentali in cui le culture usate non sono protette rendendo dunque gli esperimenti irripetibili, e nei rapporti di campo in cui non sono tenuti i campioni. Le pubblicazioni cartacee del passato hanno fornito e continuano a fornire una fonte di informazioni che possono essere sempre consultate; mettere le informazioni on line in depositi di biblioteche ufficiali come JSTOR è una salvaguardia in più.
Una revisione da esperti del settore, anche se non impermeabile, e sempre dibattuta (vedi per es., http://www.nature.com/nature/peerreview/debate/) è raccomandata come controllo di qualità prima della pubblicazione. La funzione di una revisione da parte di esperti nel caso di novità tassonomiche non è quella di esercitare una censura sulle decisioni tassonomiche ma di giudicare i nuovi Generi rispetto a questi principi ai quali la comunità micologica dovrebbe aderire.
CONCLUSIONI
Incoraggiamo tutti i micologi che intraprendono studi filogenetici a livello di Genere ad aderire a queste linee guida e inoltre invitiamo gli editori e i revisori esperti a tenerle in mente quando esaminano un lavoro per la pubblicazione.
Nel riconoscere che questi problemi sono ad ampio raggio in micologia e possono frustrare la comunicazione all’interno dell’argomento, noi lodiamo un approccio cauto all’introduzione di cambiamenti a livello generico.
Infine, accogliamo e auspichiamo valutazioni critiche delle introduzioni di nuovi taxa a tutti i livelli tassonomici, specialmente Famiglie e Ordini, in tutti i gruppi di funghi.








